När man säger att en gen ”uttrycks” eller att den ”är aktiv” betyder det att informationen överförs från DNA:t till ett budbärar-RNA (mRNA = messenger RNA) för att sedan översättas till ett protein.
DNA -> mRNA -> Protein
Francis Crick kallade detta för den centrala dogmen inom molekylärbiologin, och detta enkla 1:1:1 förhållande har utgjort grunden för i princip all molekylärbiologi ända sedan dess. Tyvärr är biologi sällan så enkelt i verkligheten, och det gäller även denna process. En gen ger nämligen oftast inte upphov till ett protein, utan till många. Och då menar jag inte bara att det bildas många likadana proteinmolekyler, utan även att en gen faktiskt kan ge upphov till flera olika sorters proteiner.
I eukaryoter och arkéer består gener av exoner och introner. Exonerna är de delar av DNA:t som innehåller själva instruktionerna för hur proteinet ska byggas. Intronerna däremot används inte för att göra proteiner utan klipps bort från mRNA-molekylerna innan dessa är färdigtillverkade. Vad intronerna har där att göra, när de ju ändå bara klipps bort, är inte helt klarlagt. Ibland kan de innehålla information som avgör när och var genen är aktiv, men oftast verkar de inte göra någonting alls.
Saken är den att när intronerna v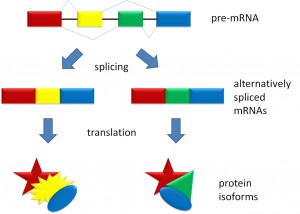 äl har klippts bort kan exonerna klistras ihop igen på flera olika sätt. Detta kallas alternativ splitsning, vilket är en försvenskning av alternative splicing, och illustreras av figuren till vänster (från Wikipedia) där boxarna i pre-mRNA:t är exoner och de tjocka strecken mellan dem är introner. Genom att sätta ihop olika kombinationer av exoner får man två olika mRNA:n och därmed två olika proteiner. Dessutom händer det ibland att inte alla introner verkligen klipps bort, vilket ger ännu fler pusselbitar att leka med. Den mer korrekta varianten av dogmen ska alltså snarare vara:
äl har klippts bort kan exonerna klistras ihop igen på flera olika sätt. Detta kallas alternativ splitsning, vilket är en försvenskning av alternative splicing, och illustreras av figuren till vänster (från Wikipedia) där boxarna i pre-mRNA:t är exoner och de tjocka strecken mellan dem är introner. Genom att sätta ihop olika kombinationer av exoner får man två olika mRNA:n och därmed två olika proteiner. Dessutom händer det ibland att inte alla introner verkligen klipps bort, vilket ger ännu fler pusselbitar att leka med. Den mer korrekta varianten av dogmen ska alltså snarare vara:
DNA -> flera olika mRNA:n -> flera olika proteiner
Hittills har forskarna generellt ganska dålig koll på vad detta alternativa splitsande egentligen har för betydelse. De flesta har istället satsat hårt de senaste åren på att mäta och jämföra hur mycket mRNA som bildas från olika gener i alla upptänkliga vävnader av alla möjliga utvecklingsstadier hos mer eller mindre alla organismer man fått tag i. Normalt skiljer forskarna då inte på olika varianter av mRNA från samma gen, utan mäter allihopa tillsammans och kallar det för ”nivån på genaktiviteten”.
Vi har vetat ett bra tag nu att alla ryggradsdjur har ganska lika arvsmassor med ungefär samma gener. Inte heller verkar det finnas några markanta skillnader i nivån på genaktiviteten mellan motsvarande organ i olika sorters ryggradsdjur. I alla fall inte tillräckligt stora skillnader för att kunna förklara varför olika ryggradsdjur faktiskt ser ganska olika ut. Men kanske har man mätt fel saker?
I en artikel som publicerades förra året i Science presenterade 17 st forskare att de upptäckt att det finns tydliga skillnader i alternativ splitsning mellan samma organ i olika ryggradsdjur. De mätte både den alternativa splitsningen och nivån på genaktiviteten i flera olika organ i människa, schimpans, orangutang, makak, mus, pungråtta, näbbdjur, kyckling, ödla och groda. Det visade sig dels att alternativ splitsning var vanligare i primater än i de övriga ryggradsdjuren och dels att den alternativa splitsningen var artberoende snarare än organberoende. Tvärtom mot nivån på genaktiviteten som var organberoende snarare än artberoende. Dessa resultat antyder att vi kanske borde ägna mer tid åt att studera alternativ splitsning och mindre tid åt att mäta mängden av olika geners mRNA om vi vill förstå vad som egentligen skiljer de olika ryggradsdjuren åt. Möjligen gäller också samma sak för växter?
Referens: Barbosa-Morais m.fl. (2012) The Evolutionary Landscape of Alternative Splicing in Vertebrate Species. Science 338, s.1587
